Lær, hvordan du bruger OCR-værktøjer, Apache Spark og andre Apache Hadoop-komponenter til at behandle PDF-billeder i skala.
Teknologier til optisk tegngenkendelse (OCR) har udviklet sig betydeligt i løbet af de sidste 20 år. Men i løbet af den tid har der været ringe eller ingen indsats for at kombinere OCR med distribuerede arkitekturer såsom Apache Hadoop for at behandle et stort antal billeder i næsten realtid.
I dette indlæg vil du lære, hvordan du bruger standard open source-værktøjer sammen med Hadoop-komponenter såsom Apache Spark, Apache Solr og Apache HBase til at gøre netop det for en informationssag om medicinsk udstyr. Specifikt vil du bruge et offentligt datasæt til at konvertere fortællende tekst til søgbare felter.
Selvom dette eksempel koncentrerer sig om information om medicinsk udstyr, kan det anvendes i mange andre scenarier, hvor behandling og vedvarende billeder er påkrævet. Forsikringsselskaber kan f.eks. gøre alle deres scannede dokumenter i skadesfiler søgbare for bedre skadeafklaring. På samme måde kunne forsyningskædeafdelingen i en produktionsfacilitet scanne alle tekniske datablade fra reservedelsleverandører og gøre dem søgbare af analytikere.
Use Case:Medicinsk enhedsregistrering
De seneste år har set en byge af ændringer inden for elektronisk lægemiddelregistrering. IDMP (Identifikation af medicinske produkter) ISO-standarden er et sådant meddelelsesformat til registrering af produkter og de stoffer, der er indeholdt i dem, hvor lægemiddel-id, emballage-id og batch-id bruges til at spore produkterne i tilfælde af uønskede oplevelser, ulovlige import, forfalskning og andre spørgsmål om lægemiddelovervågning. Standarden kræver, at ikke kun nye produkter skal registreres, men at den ældre/arkiverede arkivering af hvert produkt, som offentligheden kan blive udsat for, også skal leveres i elektronisk form.
For at overholde IDMP-standarder i forskellige virksomheder skal virksomheder være i stand til at trække og behandle data fra flere datakilder, såsom RDBMS samt, i nogle tilfælde, ældre produktdatablade. Selvom det er velkendt, hvordan man indtager data fra RDBMS via teknologier som Apache Sqoop, kræver legacy dokumentbehandling lidt mere arbejde. For det meste skal dokumenterne indlæses, og relevant tekst skal udtrækkes programmatisk i skala ved hjælp af eksisterende OCR-teknologier.
Datasæt
Vi vil bruge et datasæt fra FDA, der indeholder alle de 510(k)-ansøgninger, der nogensinde er indsendt af producenter af medicinsk udstyr siden 1976. Afsnit 510(k) i Food, Drug and Cosmetic Act kræver, at enhedsproducenter, der skal registrere sig, skal underrette FDA af deres hensigt om at markedsføre et medicinsk udstyr mindst 90 dage i forvejen.
Dette datasæt er nyttigt af flere årsager i dette tilfælde:
- Dataene er gratis og i det offentlige domæne.
- Dataene passer lige ind i den europæiske forordning, som aktiveres i juli 2016 (hvor producenterne skal overholde nye datastandarder). FDA-fyldninger har vigtige oplysninger, der er relevante for at få et komplet overblik over IDMP.
- Formatet på dokumenterne (PDF) giver os mulighed for at demonstrere enkle, men effektive OCR-teknikker, når vi håndterer dokumenter i flere formater.
For effektivt at indeksere disse data skal vi udtrække nogle felter fra billederne. Nedenfor er et eksempeldokument med de potentielle felter, der kan udtrækkes.
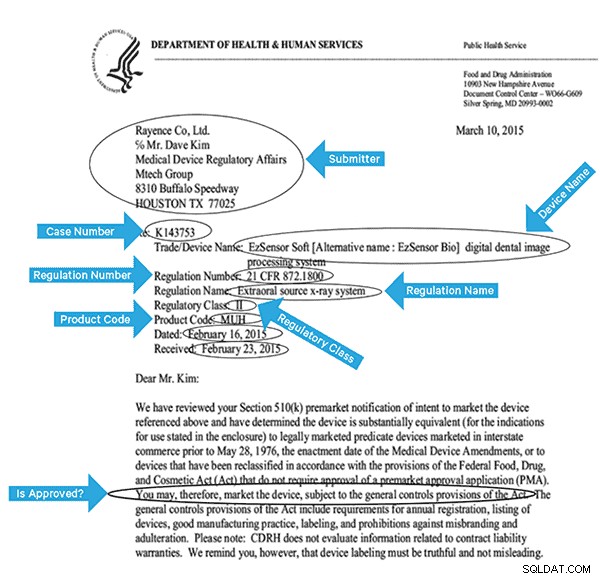
Arkitektur på højt niveau
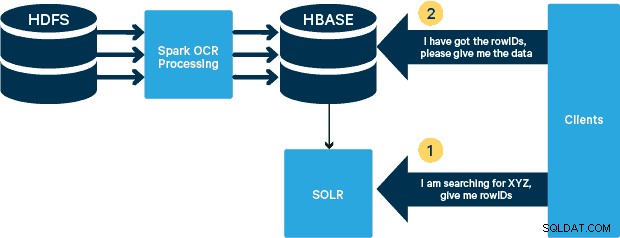
I dette tilfælde gemmes PDF'erne i HDFS og behandles ved hjælp af Spark- og OCR-biblioteker. (Indtagelsestrinnet er uden for dette indlægs omfang, men det kunne være så simpelt som at køre hdfs -dfs -put eller ved at bruge en webhdfs-grænseflade.) Spark tillader brugen af næsten identisk kode i en Spark Streaming-applikation til streaming næsten i realtid, og HBase er et perfekt lagringsmedium til tilfældig adgang med lav latens - og er velegnet til lagring af billeder, med den nye MOB-funktionalitet, til at starte op. Cloudera Search (som er bygget oven på Apache Solr) er den eneste søgeløsning, der integrerer indbygget med HBase, og derved giver dig mulighed for at bygge sekundære indekser.
Opsætning af tabellen over medicinsk udstyr i HBase
Vi vil holde skemaet til vores use case ligetil. Række-ID'et vil være filnavnet, og der vil være to kolonnefamilier:"info" og "obj". Kolonnefamilien "info" vil indeholde alle de felter, vi har udtrukket fra billederne. "obj"-kolonnefamilien vil indeholde bytes af det faktiske binære objekt, i dette tilfælde PDF. Navnet på tabellen i vores tilfælde vil være "mdds."
Vi vil drage fordel af HBase MOB (medium object) funktionalitet introduceret i HBASE-11339. For at konfigurere HBase til at håndtere MOB kræves et par ekstra trin, men bekvemt kan instruktionerne findes på dette link.
Der er mange måder at oprette tabellen i HBase på programmatisk (Java API, REST API eller en lignende metode). Her vil vi bruge HBase-skallen til at oprette "mdds"-tabellen (med vilje ved at bruge et beskrivende kolonnefamilienavn for at gøre tingene lettere at følge). Vi ønsker at få "info"-kolonnefamilien replikeret til Solr, men ikke MOB-dataene.
Kommandoen nedenfor vil oprette tabellen og aktivere replikering på en kolonnefamilie kaldet "info." Det er afgørende at angive muligheden REPLICATION_SCOPE => '1' , ellers får HBase Lily Indexer ingen opdateringer fra HBase. Vi ønsker at bruge MOB-stien i HBase til objekter større end 10 MB. For at opnå det opretter vi også en anden kolonnefamilie, kaldet "obj", ved hjælp af følgende parametre for MOB'er:
IS_MOB => sand, MOB_THRESHOLD => 10240000
IS_MOB parameter angiver, om denne kolonnefamilie kan gemme MOB'er, mens MOB_THRESHOLD angiver efter hvor stort objektet skal være for at det kan betragtes som en MOB. Så lad os oprette tabellen:
opret 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => sand, MOB_THRESHOLD => 10240000} For at bekræfte, at tabellen blev oprettet korrekt, skal du køre følgende kommando i HBase-shell:
hbase(main):001:0> beskriv 'mdds'Tabel mdds er ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', => '1CO' , VERSIONS => '1', KOMPRESSION => 'INGEN', MIN_VERSIONS => '0', TTL => 'FOR ALLTID', KEEP_DELETED_CELLS => 'FALSK', BLOCKSIZE => '65536', IN_MEMORY => 'falsk', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'INGEN', BLOOMFILTER => 'RÆKKE', REPLICATION_SCOPE => '0', KOMPRESSION => 'INGEN', VERSIONER => '1', MIN_VERSIONS => '0', TTL => 'FOR ALLTID', MOB_THRESHOLD => '10240000', IS_MOB => 'sand', KEEP_DELETED_CELLS => 'FALSK', BLOCKSIZE => '65536', IN_MEMORY' => 'falsk => 'true'}2 række(r) på 0,3440 sekunder Behandling af scannede billeder med Tesseract
OCR er nået langt med hensyn til at håndtere skrifttypevariationer, billedstøj og problemer med justeringer. Her vil vi bruge open source OCR-motoren Tesseract, som oprindeligt blev udviklet som proprietær software på HP-laboratorier. Tesseract-udvikling er siden blevet frigivet som en open source-software og været sponsoreret af Google siden 2006.
Tesseract er et meget bærbart softwarebibliotek. Den bruger Leptonica billedbehandlingsbiblioteket til at generere et binært billede ved at lave adaptiv tærskelværdi på et gråt eller farvet billede.
Forarbejdning følger en traditionel trin-for-trin pipeline. Følgende er den grove flow af trin:
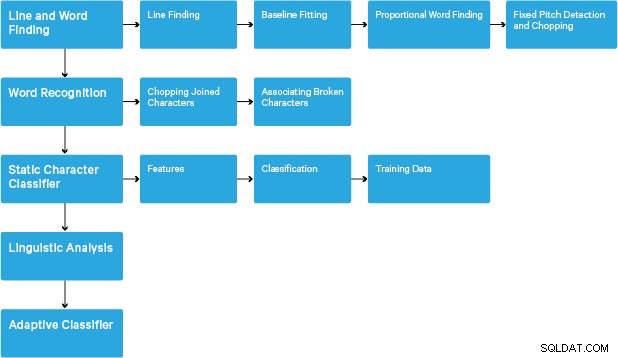
Behandlingen starter med en forbundet komponentanalyse, som resulterer i lagring af de fundne komponenter. Dette trin hjælper med inspektion af indlejring af konturer og antallet af børne- og barnebarnskonturer.
På dette trin samles konturerne, udelukkende ved at indlejre, til binære store objekter (BLOB'er). BLOB'er er organiseret i tekstlinjer, og linjerne og områderne analyseres for fast pitch eller proportional tekst. Tekstlinjer er opdelt i ord forskelligt afhængigt af typen af tegnafstand. Tekst med fast tonehøjde hugges straks af karakterceller. Proportional tekst er opdelt i ord ved hjælp af bestemte mellemrum og uklare mellemrum.
Anerkendelsen fortsætter derefter som en to-pass proces. I det første gennemløb forsøger man at genkende hvert ord efter tur. Hvert ord, der er tilfredsstillende, videregives til en adaptiv klassifikator som træningsdata. Den adaptive klassifikator får så en chance for mere præcist at genkende tekst længere nede på siden. Da den adaptive klassifikator måske har lært noget nyttigt for sent til at give et bidrag nær toppen af siden, køres der en anden omgang over siden, hvor ord, der ikke blev genkendt godt nok, genkendes igen. En sidste fase løser uklare mellemrum og kontrollerer alternative hypoteser for x-højden for at lokalisere tekst med små bogstaver.
Tesseract i sin nuværende form er fuldt unicode-kompatibel og trænet til flere sprog. Baseret på vores forskning er det et af de mest nøjagtige open source-biblioteker til rådighed for OCR. Som tidligere nævnt bruger Tesseract Leptonica. Vi gør også brug af Ghostscript til at opdele PDF-filerne i billeder. (Du kan opdele i billedkomprimeringsformat efter eget valg; vi valgte PNG.) Disse tre biblioteker er skrevet i C++, og for at kalde dem fra Java/Scala-programmer, skal vi bruge implementeringer af tilsvarende Java Native Interfaces. I vores arbejde bruger vi JNI-bindingerne fra JavaPresets. (Byggevejledningen kan findes nedenfor.) Vi brugte Scala til at skrive Spark-driveren.
val renderer :SimpleRenderer =ny SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica læser de opdelte billeder fra det forrige trin ind.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.Stream(.rayByteAram) ) ).capacity( ))
Vi bruger derefter Tesseract API-kald til at udtrække teksten. Vi antager, at dokumenterne er på engelsk her, derfor er den anden parameter til Init-metoden "eng."
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Efter at billederne er behandlet, udtrækker vi nogle felter fra teksten og sender dem til HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** Konfigurer og åbn en HBase-forbindelse */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( filnavn )) /** * Udtræk felter her ved hjælp af regexes * Opret Put-objekter og send til HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. linjer matcher { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( adr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "matchede ikke et regex") } ……. lines.split("\n").foreach { val regNumRegex ="""Regulationsnummer:\s+(.+)""".r val regNameRegex ="""Regulationsnavn:\s+(.+)""" .r …….. ……. _ match { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "") } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….} Hvis du ser nærmere på koden ovenfor, lige før vi sender Put-objektet til HBase, indsætter vi de rå PDF-bytes i "obj"-kolonnefamilien i tabellen. Vi bruger HBase som et lagerlag for de udtrukne felter samt råbilledet. Dette gør det hurtigt og bekvemt for applikationen at udtrække det originale billede, hvis det er nødvendigt. Den fulde kode kan findes her. (Det er værd at bemærke, at mens vi brugte standard HBase API'er til at skabe Put-objekter til HBase, i et rigtigt produktionssystem, ville det være klogt at overveje at bruge SparkOnHBase API'er, som giver mulighed for batchopdateringer til HBase fra Spark RDD'er.)
Execution Pipeline
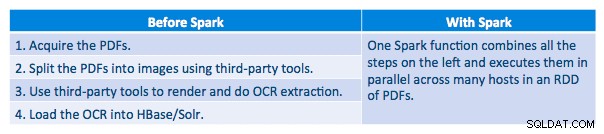
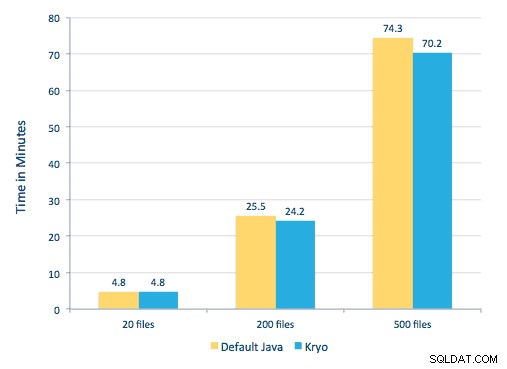
Vi var i stand til at behandle hver PDF i en seriel ramme. For at skalere behandlingen valgte vi at behandle disse PDF'er på en distribueret måde ved hjælp af Spark. Følgende diagram viser, hvordan vi kombinerer forskellige stadier af denne behandling for at omdanne arbejdsgangen til et simpelt makrokald fra Spark og få dataene indlæst i HBase.
Vi forsøgte også at foretage en sammenligning mellem serialiseringsmetoder, men med vores datasæt så vi ikke en signifikant forskel i ydeevne.
Opsætning af miljø
Anvendt hardware:Fem-node-klynge med 15 GB hukommelse, 4 vCPU'er og 2x40 GB SSD
Da vi brugte C++-biblioteker til behandling, brugte vi JNI-bindingerne, som kan findes her.
Byg JNI-bindingerne til Tesseract og Leptonica fra javaCPP-forudindstillinger:
-
- På alle noder:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Byg Leptonica.
cd leptonica./cppbuild.sh installer leptonicacd cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Byg Tesseract.
- På alle noder:
cd tesseract./cppbuild.sh installer tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- Byg javaCPP-forudindstillinger.
mvn clean install --projects leptonica,tesseract
Vi bruger Ghostscript til at udtrække billederne fra PDF'erne. Instruktioner til at bygge Ghostscript, svarende til versionerne af Tesseract og Leptonica, der bruges her, er som følger. (Sørg for, at Ghostscript ikke er installeret i systemet via pakkehåndteringen.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo lav &&gør soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(Afhænger af din ldpath indstilling, skal du muligvis gøre):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Sørg for, at alle de nødvendige biblioteker er i klassestien. Vi placerer alle de relevante krukker i en mappe kaldet lib. Komma er vigtigt nedenfor:
$ for i i `ls lib/*`; eksporter MY_JARS=./$i,$MY_JARS; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Vi påberåber os Spark-programmet som følger. Vi skal specificere extraLibraryPath for native Ghostscript-biblioteker; den anden conf er nødvendig for Tesseract.
spark-submit --jars $MY_JARS --antal-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Oprettelse af en Solr-samling
Solr integrerer ganske problemfrit med HBase via Lily HBase Indexer. For at forstå, hvordan integrationen af Lily Indexer-integration med HBase foregår, kan du friske op via vores tidligere indlæg i afsnittet "Forstå HBase-replikering og Lily HBase Indexer".
Nedenfor skitserer vi de trin, der skal udføres for at oprette indekserne:
- Generer et eksempel på en schema.xml-konfigurationsfil:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Rediger filen schema.xml i
$HOME/solrcfg, med angivelse af de felter, vi skal bruge til vores samling. Den fulde fil kan findes her. - Upload Solr-konfigurationerne til ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Generer Solr-samlingen med 2 shards (-s 2) og 2 replikaer (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
I kommandoen ovenfor oprettede vi en Solr-samling med to shards (-s 2) og to replikaer (-r 2) parametre. Parametrene var tilstrækkelige for vores korpus, men i en egentlig udrulning ville man skulle indstille antallet baseret på andre overvejelser uden for vores diskussionsområde her.
Registrering af Indexer
Dette trin er nødvendigt for at tilføje og konfigurere indekseren og HBase-replikeringen. Kommandoen nedenfor vil opdatere ZooKeeper og tilføje mdds_indexer som en replikeringspeer for HBase. Det vil også indsætte konfigurationer i ZooKeeper, som Lily HBase Indexer vil bruge til at pege på den rigtige samling i Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumenter:
-n mdds_indexer– angiver navnet på den indeksering, der vil blive registreret i ZooKeeper-c indexer-config.xml– konfigurationsfil, der specificerer indekseringsadfærd-cp solr.zk=localhost:2181/solr– angiver placeringen af ZooKeeper og Solr config. Dette bør opdateres med den miljøspecifikke placering af ZooKeeper.-cp solr.collection=mdds_collection– angiver, hvilken samling der skal opdateres. Husk Solr-konfigurationstrinnet, hvor vi oprettede samling1.
index-config.xml fil er relativt ligetil i dette tilfælde; alt det gør, er at specificere for indekseren, hvilken tabel der skal ses på, den klasse, der skal bruges som mapper (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ), og placeringen af Morphline-konfigurationsfilen. Som standard er mapping-type indstillet til række , i hvilket tilfælde Solr-dokumentet bliver den fulde række. Param name="morphlineFile" angiver placeringen af Morphlines-konfigurationsfilen. Placeringen kunne være en absolut sti til din Morphlines-fil, men da du bruger Cloudera Manager, skal du angive den relative sti som morphlines.conf.
Indholdet af hbase-indexer-konfigurationsfilen kan findes her.
Konfiguration og start af Lily HBase Indexer
Når du aktiverer Lily HBase Indexer, skal du angive Morphlines-transformationslogikken, der vil tillade denne indekser at parse opdateringer til tabellen Medical Device og udtrække alle relevante felter. Gå til Services og vælg Lily HBase Indexer, som du tidligere tilføjede. Vælg Konfigurationer->Vis og rediger->Service-Wide->Morphlines . Kopiér og indsæt Morphlines-filen.
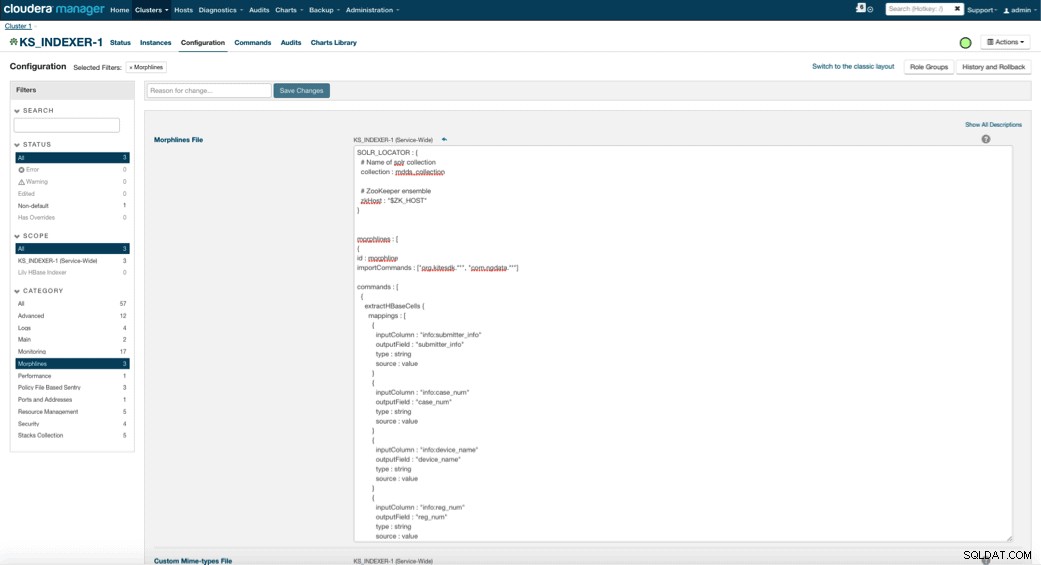
Det medicinske udstyrs morphlinesbibliotek udfører følgende handlinger:
- Læs HBase-e-mail-begivenhederne med
extractHBaseCellskommando - Konverter dato/tidsstempler til et felt, som Solr vil forstå, med
convertTimestampkommandoer - Slet alle de ekstra felter, som vi ikke har angivet i schema.xml, med
sanitizeUknownSolrFieldskommando
Download en kopi af denne Morphlines-fil herfra.
En vigtig bemærkning er, at id-feltet bliver automatisk genereret af Lily HBase Indexer. Denne indstilling kan konfigureres i index-config.xml-filen ovenfor ved at angive unikke-nøgle-felt-attributten. Det er en god praksis at forlade standardnavnet på id'et – da det ikke var angivet i xml-filen ovenfor, blev standard-id-feltet genereret og vil være en kombination af RowID.
Adgang til dataene
Du har valget mellem mange visuelle værktøjer for at få adgang til de indekserede billeder. HUE og Solr GUI er begge meget gode muligheder. HBase muliggør også en række adgangsteknikker, ikke kun fra en GUI, men også via HBase-shell, API og endda simple scriptteknikker.
Integration med Solr giver dig stor fleksibilitet og kan også give meget enkle såvel som avancerede søgemuligheder for dine data. Konfiguration af Solr schema.xml-filen, således at alle felter inden for e-mail-objektet er gemt i Solr, giver brugerne adgang til hele meddelelsesteksterne via en simpel søgning, med en afvejning af lagerplads og computerkompleksitet. Alternativt kan du konfigurere Solr til kun at gemme et begrænset antal felter, såsom id'et. Med disse elementer kan brugere hurtigt søge i Solr og hente rowID, som igen kan bruges til at hente individuelle felter eller hele billedet fra selve HBase.
Eksemplet ovenfor gemmer kun række-ID'et i Solr, men indekserer på alle felter udtrukket fra billedet. Søgning i Solr i dette scenarie henter HBase række-id'er, som du derefter kan bruge til at forespørge HBase. Denne type opsætning er ideel til Solr, da den holder lageromkostningerne lave og udnytter Solrs indekseringsmuligheder fuldt ud.
Eksempelforespørgsler
Nedenfor er nogle eksempler på forespørgsler, der kan udføres fra applikationen til Solr. Ideen er, at klienten i første omgang vil forespørge på Solr-indekser og returnere rowID'et fra HBase. Forespørg derefter HBase for resten af felterne og/eller det originale råbillede.
- Giv mig alle dokumenter, der blev arkiveret mellem følgende datoer:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59.999Z TIL 2010-02-06T23:59:59.999Z]
- Giv mig dokumenter, som er arkiveret under det lovpligtige navn for mobile røntgensystemer:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile røntgensystem
- Giv mig alle dokumenter, der er indgivet fra kinesiske producenter:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Id'erne fra Solr-dokumenter er række-id'erne i HBase; den anden del af forespørgslen vil gå til HBase for at udtrække dataene (inklusive den rå PDF, hvis det kræves).
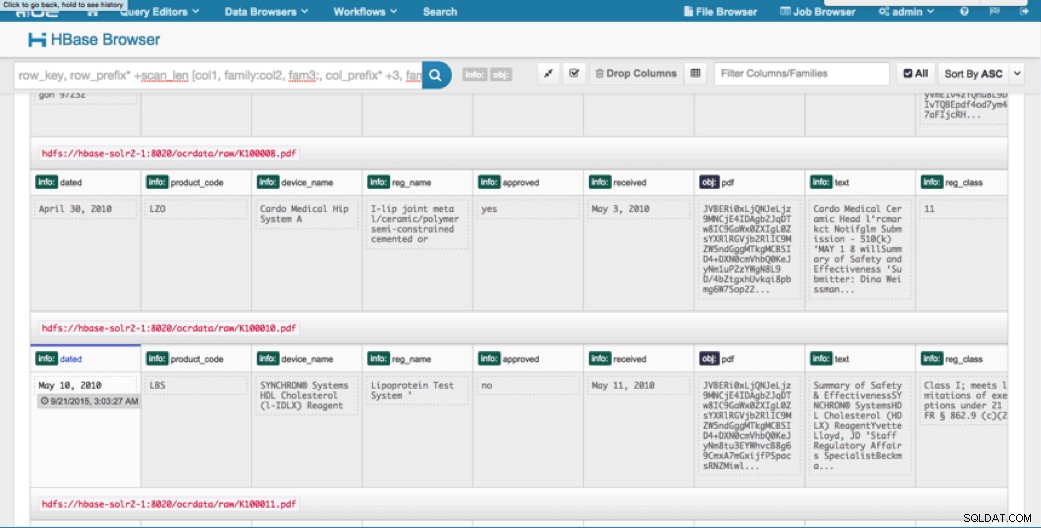
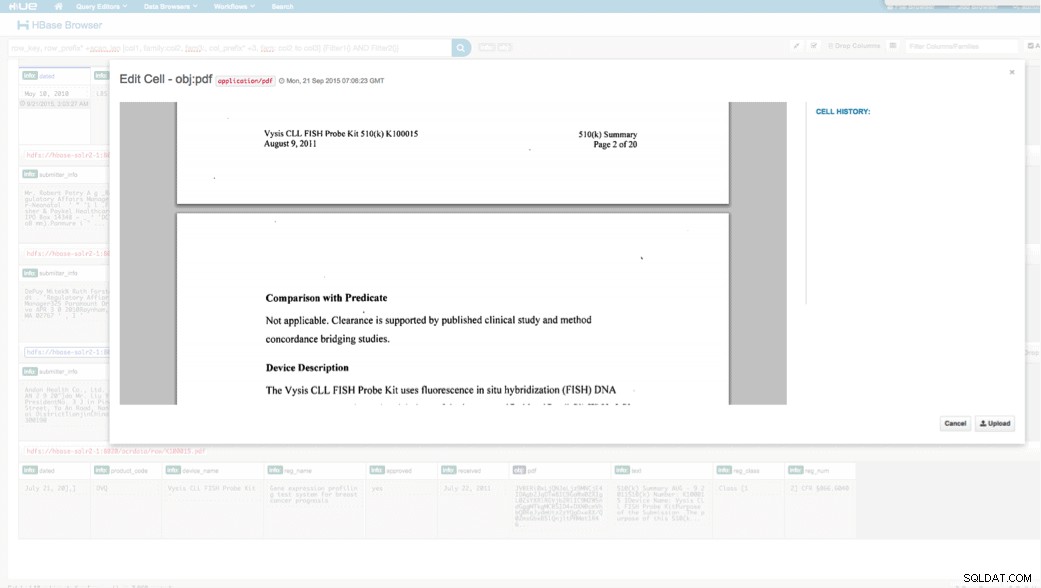
Adgang via HUE
Vi kan se de uploadede data via HBase Browser i HUE. En stor ting ved HUE er, at den kan registrere binære filer til PDF og gengive dem, når der klikkes på dem.
Nedenfor er et snapshot af visningen af de parsede felter i HBase-rækker og også en gengivet visning af et af PDF-objekterne gemt som en MOB i obj-kolonnefamilien.
Konklusion
I dette indlæg har vi demonstreret, hvordan man bruger standard open source-teknologier til at udføre OCR på scannede dokumenter ved hjælp af et skalerbart Spark-program, lagring i HBase for hurtig hentning og indeksering af de udtrukne oplysninger i Solr. Det skal være tydeligt, at:
- I betragtning af meddelelsesspecifikationens format kan vi udtrække felter og værdipar og gøre dem søgbare via Solr.
- Disse felter fra data kan opfylde IDMP-kravene til at gøre de ældre data elektroniske, hvilket træder i kraft engang næste år.
- Felterne såvel som råbilleder kan bevares i HBase og tilgås via standard API'er.
Hvis du har brug for at behandle scannede dokumenter og kombinere dataene med forskellige andre kilder i din virksomhed, kan du overveje at bruge en kombination af Spark, HBase, Solr sammen med Tesseract og Leptonica. Det kan spare dig for en betydelig mængde tid og penge!
Jeff Shmain er Senior Solution Architect hos Cloudera. Han har mere end 16 års erfaring i den finansielle branche med stærk forståelse for handel med sikkerhed, risici og regler. I løbet af de sidste par år har han arbejdet på forskellige use case-implementeringer hos 8 ud af 10 verdens største investeringsbanker.
Vartika Singh er Senior Solution Consultant hos Cloudera. Hun har over 12 års erfaring med anvendt maskinlæring og softwareudvikling.
